MST-Analysis
蛋白质或者小分子在生物液体(例如血清或细胞裂解物)中会发生相互作用,可以把这种作用想象成你穿着毛衣经过长满苍耳的灌木丛时,那些满是钩子的种子粘的你满身都是,但是如果你穿的是冲锋衣,那么可能就一颗也粘不上。这些分子化合物之间也是如此,它们之间有选择性地相互结合。了解这些结合的亲和度是研发新药的关键参考。
把一堆宏观上看不到的蛋白质放入纤细透光的毛细管中,再加热到适合的温度(差不多是人的体温,因为这些小东西对于温度很挑剔),就可能看到在这种自由、接近人体内环境的场景中,分子会相互结合。如果它们相互结合了,那么对光的折射反射率就不同了,所以在宏观上用荧光强度就可以从侧面反映它们结合的强度 1。另外,两种分子之间的结合比例到底是多少也是一个关键问题,所以需要配比不同的浓度梯度的样品都测测看。
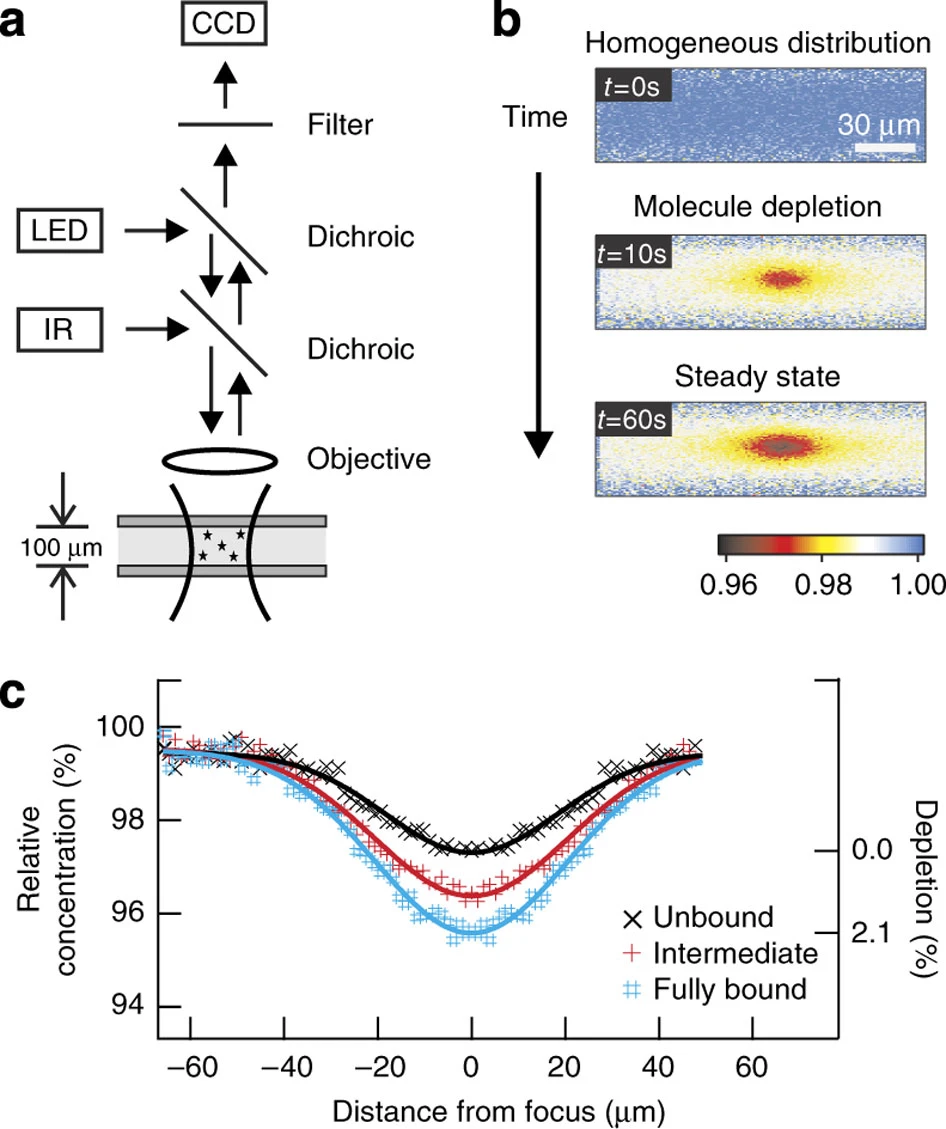
a. 观测这个微观过程的设备结构,b. 刚开始因为温度不合适,分子之间不结合,加热之后它们就快速行动起来,并呈现出宏观的变化,c. 从侧面的荧光强度曲线可以观测到它们的结合强度
理论背景
当溶液加热时,分子就会沿着温度梯度移动(详见热致扩散),移动方向和质量扩散方向相反。这两种相反的浓度变化趋势会形成一种平衡:
\begin{equation} \frac{c}{c_{0}}=\exp[-S_{T} \cdot(T-T_{0})] \end{equation}
这里的相对浓度取决于Soret系数\(S_{T}\)和温度梯度\(\Delta T=T-T_{0}\),其中,Soret系数的确定还需要考虑很多物理现象,包括表面参数(电荷、疏水性)、缓冲液中离子移动产生的电场等。不过,这里直接给他套个简单模型差也差不多了:
\begin{equation} S_{T}=\frac{A}{kT}\left( -\Delta s_{\rm hyd}(T)+\frac{\beta \sigma^{2}_{\rm eff}}{4\varepsilon \varepsilon_{0}T} \cdot \lambda_{\rm DH} \right) \end{equation}
\(A\)是分子的表面积,\(\sigma_{\rm eff}\)表示有效电荷,\(\Delta s_{\rm hyd}\)表示分子-溶液界面的水合熵,\(\lambda_{\rm DH}\)表示德拜-休克尔筛分长度,\(\varepsilon\)表示介电常数,\(\beta\)表示其温度导数。
当蛋白质和分子相互作用时,表面积\(A\)会改变,\(\Delta s_{\rm hyd}\)水合熵会变,有效电荷\(\sigma_{\rm eff}\)也会变。即一点点微小的构型改变,我们都可以通过宏观的统计数据(例如浓度)捕捉到。
所以,基于物质作用定律可以推导出如下的经验公式:
\begin{equation} \frac{BL}{B_{0}}=\frac{(L_{0}+B_{0}+K_{d})-\sqrt{(L_{0}+B_{0}+K_{d})^{2}-4L_{0}B_{0}}}{2B_{0}} \end{equation}
这里 \(B\) 是指结合位点,\(L\)是指配体,\(BL\)是它们形成的复合物的浓度。\(L_{0}\)表示每个数据点添加配体的量,\(B_{0}\)是指结合状态对应的总浓度。\(K_{d}\)是解离常数,它量化了配体与特定受体之间的亲和程度。
基于一种线性变化的思路,你的配体浓度低了,那么溶液中只有部分受体处于结合状态,你只有滴滴滴滴到差不多过量了以后,才能保证所有的受体都和配体结合了。那么那个就是饱和浓度,但是我们刚开始怎么会知道那东西是多少,所以你就得用各种浓度梯度去试出来。这样,我们就可以写出一条随浓度连续变换的插值关系来:
\begin{equation} \Delta c_{\rm measured}=f\cdot \Delta c_{\rm bound}+(1-f)\cdot \Delta c_{\rm unbound} \end{equation}
数据分析
前面已经说了,浓度是怎么变,荧光强度就怎么变,所以用荧光强度值\(F\)代入就有:
\begin{equation} F=(1-f(c))F_{\rm unbound} + f(c)F_{\rm bound} \end{equation}
注意:这里的 \(F_{\rm unbound}\)和\(F_{\rm bound}\)是不知道的,只能按照下面这个公式去代入数值去估计:
\begin{equation} f(c)=\frac{c+c_T+K_d-\sqrt{\left(c+c_{T} + K_d\right)^2+4 c c_T}}{2 c_T} \end{equation}
以上带入的参数都应该首先进行归一化,\(\Delta c_{\rm measured}\)就对应于\(\frac{BL}{B_{0}}\),也对应于归一化的荧光强度 \(F_{\rm norm}\)。其中,荧光的强度的归一化一般遵照一条约定俗成的规则如下图所示:
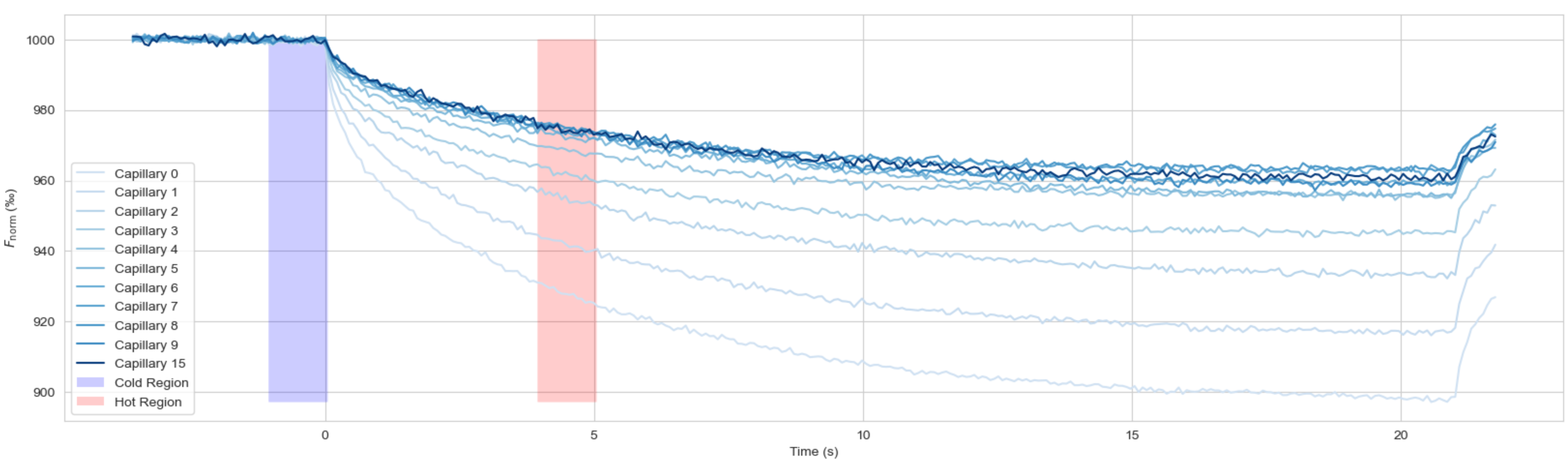
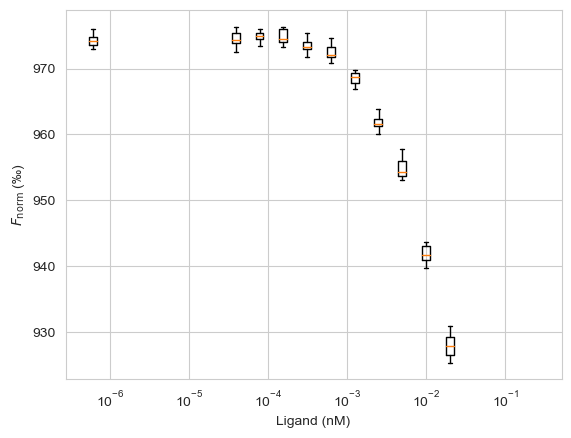
蓝色区域一般作为归一化常数\(F_{0}\),对应于加热前荧光强度,红色区域作为被归一化荧光强度\(F_{1}\)。之所以这么选择是因为:在红外激光加热几秒钟后,即使扩散缓慢且在测量时间内没有达到稳态,浓度曲线通常在几秒钟后即可区分。要的就是这种区分度,所以在一般情况下取其第4秒到第5秒这一秒内的荧光强度样本点就可以了,这只是一种习惯。每个箱线图都对应于一组一秒内的荧光强度,绝对数值不那么重要,数值随配体的浓度变化规律才重要。
Python 拟合
首先导入必要的库,一般仪器导出的 .moc 文件就是个 SQLite 文件,已经有雷锋写好了解码函数2可以用于解码,导入这个 MSTProcess 即可:
import numpy as np
import MSTProcess as MST
from scipy.optimize import curve_fit, minimize
# 导入MST实验数据
MOCfile = MST.openMOCFile('../data/xxxx.moc')
然后定义结合比例公式:
def K_d_model(c, K_d, F_unbound, F_bound, c_T):
def func(c, K_d):
# Protein-binding assays in biological liquids using microscale thermophoresis
val = (c + c_T + K_d - np.sqrt((c + c_T + K_d) ** 2 - 4 * c * c_T)) / (2 * c_T)
return val
return (1 - func(c, K_d)) * F_unbound + func(c, K_d) * F_bound
接着定义模型拟合的损失函数:
def robust_loss(params, c_T, c, y_data):
K_d, F_unbound, F_bound = params
predictions = K_d_model(c, K_d, F_unbound, F_bound, c_T)
residuals = predictions - y_data
loss = np.sum(2 * (np.sqrt(1 + (residuals ** 2))) - 2)
# loss = np.sum(residuals ** 2)
return loss
然后进行拟合:
# 浓度序列
x_data = concentr_list
# 归一化后的荧光强度序列
y_data = np.asarray(F1_list.mean(axis=1))
# 初始的猜测值 [K_d, F_unbound, F_bound] 用于帮助更快求解
init_params = [0.1, 974, 929]
res = minimize(robust_loss, init_params, args=(c_T, x_data, y_data))
# 打印拟合结果
print(f"K_d: {res.x[0]}, F_unbound: {res.x[1]}, F_bound: {res.x[2]}")
完整实现代码参见:https://github.com/lion-forest/MST-anaylysis
名词解释3
- 热致扩散,Thermaldiffusivity:质扩散和热传导耦合引起的热质扩散,常称之为索瑞效应和杜伏效应。在热质扩散中,由于温度梯度引起混合物中出现附加的浓度梯度而造成传质,是于1893年由索瑞(C. Soret)在液体中发现的。杜伏效应则是指由于各种物质互相扩散引起温度梯度,或者说,由于有浓度梯度引起温度梯度,这是杜伏(L. Dufour)于1872年在气体中发现的。杜伏效应是索瑞效应的逆效应。
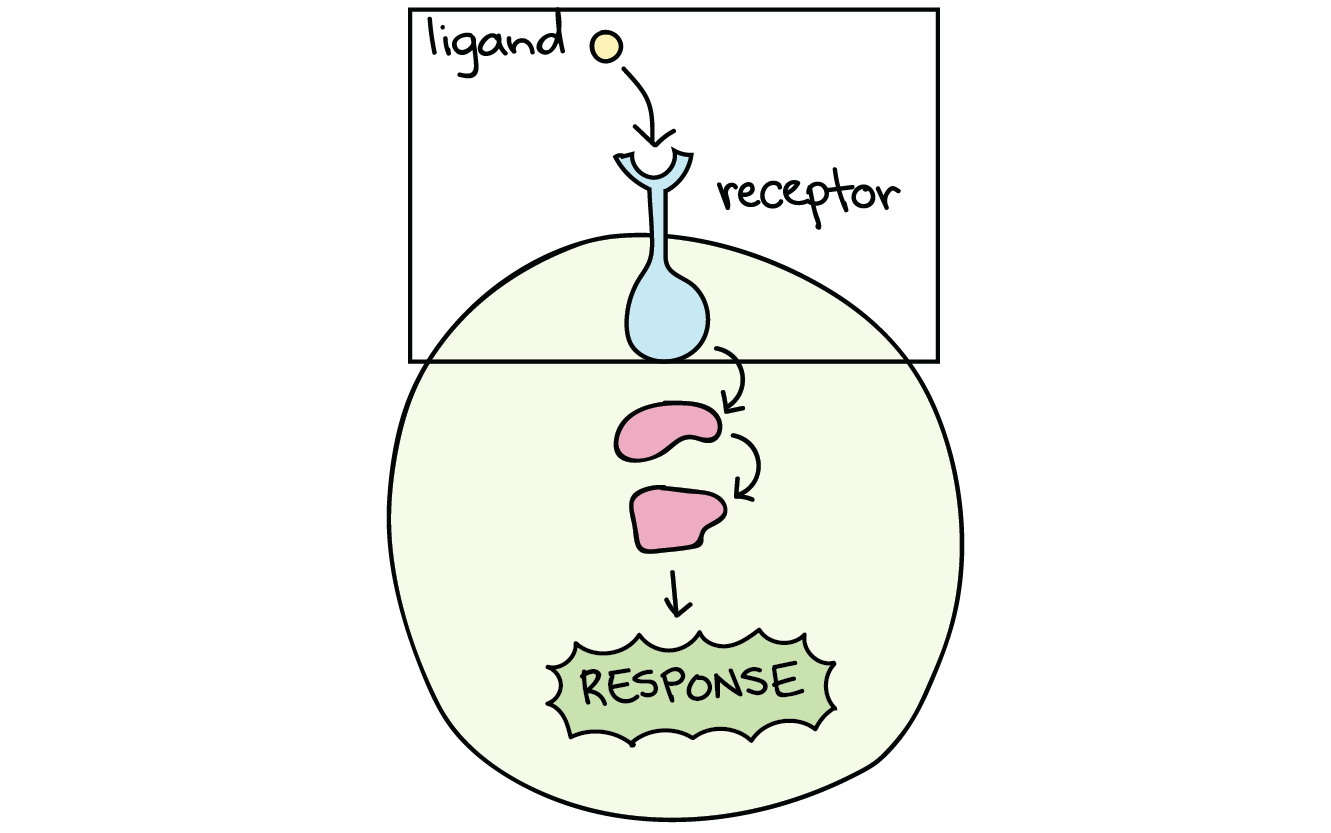
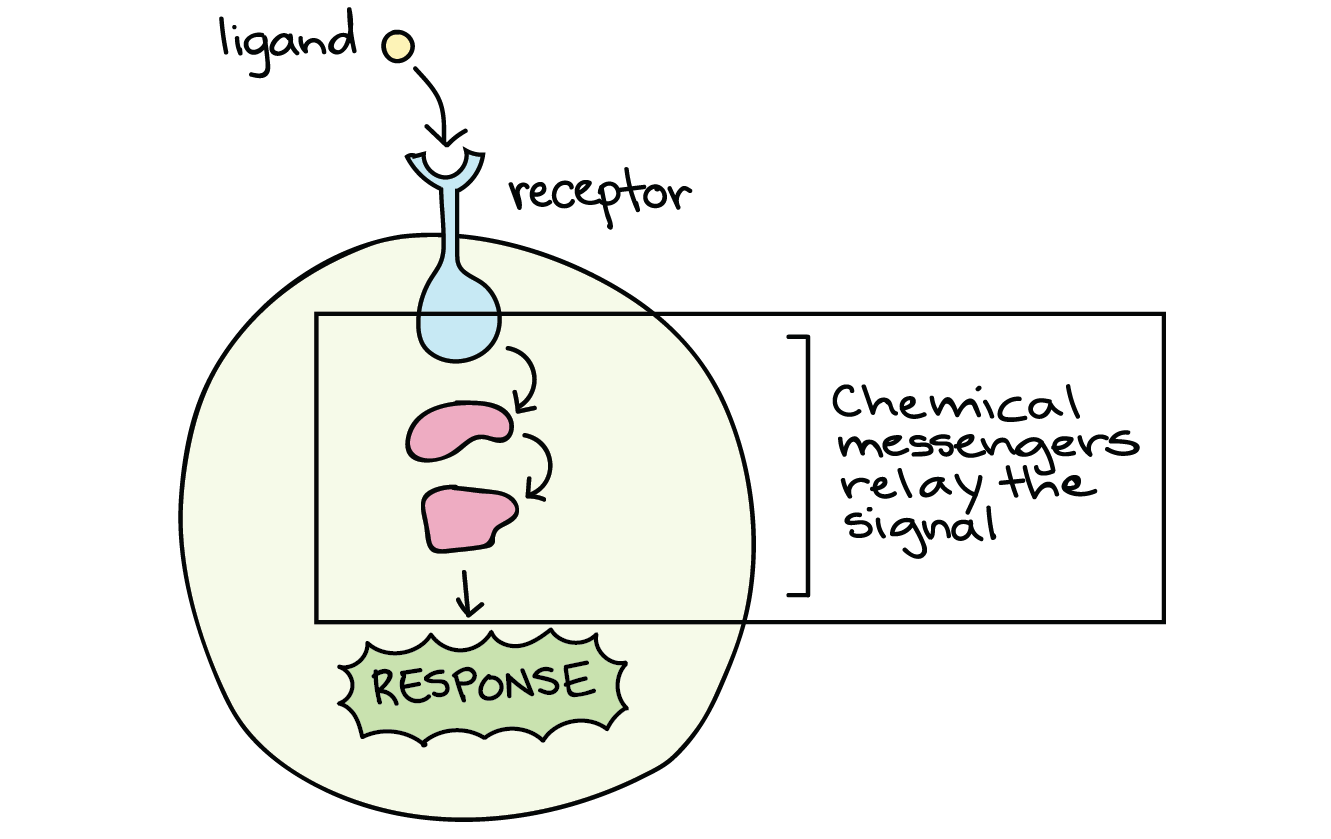
- 受体(Receptor):指一类能传导细胞外信号,并在细胞内产生特定效应的分子。包括膜受体和胞内受体。
- 配体(Ligand):指一种能与受体结合以产生某种生理效果的物质。细胞外能与受体结合的分子一般称之为配体,包括激素生长因子,细胞因子,神经递质,还有其他各种各样的小分子化合物
-
Wienken, C., Baaske, P., Rothbauer, U. et al. Protein-binding assays in biological liquids using microscale thermophoresis. Nat Commun 1, 100 (2010). https://doi.org/10.1038/ncomms1093 ↩
-
https://zh.khanacademy.org/science/biology/cell-signaling/mechanisms-of-cell-signaling/a/introduction-to-cell-signaling ↩
Enjoy Reading This Article?
Here are some more articles you might like to read next: